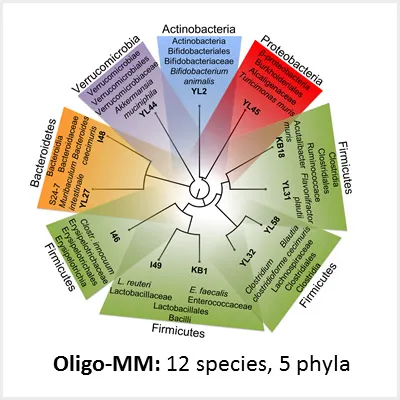
OMM12: A synthetic bacterial community
Eine zentrale Herausforderung bei der Erforschung des Darmmikrobioms besteht darin zu verstehen, wie die Interaktionen zwischen den einzelnen Mikroorganismen den Aufbau, die Dynamik und die Funktionalität der Gemeinschaft beeinflussen. Gnotobiotische Mäuse, die mit definierten mikrobiellen Gemeinschaften besiedelt sind, sind zu einem wichtigen Werkzeug geworden, um diese Frage zu beantworten. Während in den meisten Studien Bakterien aus dem Menschen verwendet werden, haben wir eine Modellgemeinschaft aus zwölf phylogenetisch unterschiedlichen Bakterien entwickelt, die aus Mäusen isoliert wurden (Brugiroux et al., 2016) (Abb. 3). Darüber hinaus haben wir eine umfassende Reihe von Protokollen und Analysewerkzeugen entwickelt, die es uns ermöglichen, diese Gemeinschaft in ihrem natürlichen Mäusewirt zu untersuchen. Diese Oligo-Maus-Mikrobiota (OMM12) ist einzigartig, weil sie zentrale physiologische und funktionelle Merkmale eines komplexen Mausmikrobioms rekapituliert. Außerdem ist die OMM12 in gnotobiotischen Mäusen langzeitstabil und ihre Zusammensetzung ist in verschiedenen Tiereinrichtungen reproduzierbar. Daher wird dieses Modell von einer wachsenden Zahl von Forschungsgruppen weltweit verwendet.
Um einen ökologischen Kontext zu schaffen, haben wir das Interaktionsnetzwerk und die metabolischen Fähigkeiten dieser synthetischen Bakteriengemeinschaft eingehend untersucht, beides Faktoren, die den Aufbau, die Dynamik und die Funktionalität der Gemeinschaft bestimmen (Weiss et al., ISME 2021). Wir verwendeten einen Bottom-up-Ansatz, der die Ergebnisse von mono- und paarweisen Co-Kultur-Experimenten mit den Beobachtungen komplexer Gemeinschaften in In-vitro-Batch-Kulturen verband. Darüber hinaus kombinierten wir die Metabolomanalyse von verbrauchten Kulturüberständen mit der genominformierten Rekonstruktion von Stoffwechselwegen und erstellten Stoffwechselmodelle des OMM12-Konsortiums. Insgesamt wurden durch diese Arbeit wichtige Interaktionsmuster zwischen den OMM12-Stämmen identifiziert, die für den Aufbau und die Funktionalität der Gemeinschaft relevant sind.
Publikations:
Brugiroux, S., Beutler, M., Pfann, C., Garzetti, D., Ruscheweyh, H.J., Ring, D., Diehl, M., Herp, S., Lotscher, Y., Hussain, S., et al. (2016). Genome-guided design of a defined mouse microbiota that confers colonization resistance against Salmonella enterica serovar Typhimurium. Nat Microbiol. 2, 16215. DOI: 10.1038/nmicrobiol.2016.215.
Eberl C, Ring D, Münch PC, Beutler M, Basic M, Slack EC, Schwarzer M, Srutkova D, Lange A, Frick JS, Bleich A, Stecher B (2020). Reproducible Colonization of Germ-Free Mice With the Oligo-Mouse-Microbiota in Different Animal Facilities. Front Microbiol. DOI: 10.3389/fmicb.2019.02999
Weiss, A.S., Burrichter, A., Durai Raj A.C., von Strempel A., Meng, C., Kleigrewe K., Münch P.C., Rössler L., Huber C., Eisenreich W., Jochum L.M., Göing S., Jung K., Lincetto C., Hübner J., Marinos G., Zimmermann, J., Kaleta C., Sanchez A., Stecher B (2021). In vitro interaction network of a synthetic gut bacterial community. ISME . DOI: 10.1038/s41396-021-01153-z